Noter
Cliquez ici pour télécharger l'exemple de code complet
Tutoriel d'images #
Un petit tutoriel sur le traçage d'images avec Matplotlib.
Commandes de démarrage #
Tout d'abord, commençons IPython. Il s'agit d'une excellente amélioration de l'invite Python standard, et elle s'intègre particulièrement bien à Matplotlib. Démarrez IPython soit directement sur un shell, soit avec le bloc-notes Jupyter (où IPython est un noyau en cours d'exécution).
Avec IPython démarré, nous devons maintenant nous connecter à une boucle d'événements GUI. Cela indique à IPython où (et comment) afficher les tracés. Pour vous connecter à une boucle GUI, exécutez la magie %matplotlib à votre invite IPython. Il y a plus de détails sur ce que cela fait exactement dans la documentation d'IPython sur les boucles d'événements GUI .
Si vous utilisez Jupyter Notebook, les mêmes commandes sont disponibles, mais les gens utilisent généralement un argument spécifique pour la magie %matplotlib :
In [1]: %matplotlib inline
Cela active le traçage en ligne, où les graphiques de tracé apparaîtront dans votre bloc-notes. Cela a des implications importantes pour l'interactivité. Pour le traçage en ligne, les commandes dans les cellules sous la cellule qui génère un tracé n'affecteront pas le tracé. Par exemple, la modification de la palette de couleurs n'est pas possible à partir des cellules situées sous la cellule qui crée un tracé. Cependant, pour d'autres backends, tels que Qt, qui ouvrent une fenêtre séparée, les cellules en dessous de celles qui créent le tracé modifieront le tracé - c'est un objet vivant en mémoire.
Ce tutoriel utilisera l'interface de traçage implicite de Matplotlib, pyplot. Cette interface maintient l'état global et est très utile pour expérimenter rapidement et facilement divers paramètres de tracé. L'alternative est l'explicite, qui convient mieux au développement d'applications volumineuses. Pour une explication des compromis entre les interfaces implicites et explicites, consultez les interfaces d' application Matplotlib (API) et le guide de démarrage rapide pour commencer à utiliser l'interface explicite. Pour l'instant, passons à l'approche implicite :
import matplotlib.pyplot as plt
import matplotlib.image as mpimg
Importation de données d'image dans des tableaux Numpy #
Matplotlib s'appuie sur la bibliothèque Pillow pour charger les données d'image.
Voici l'image avec laquelle nous allons jouer :

C'est une image PNG RVB 24 bits (8 bits pour chacun des R, G, B). Selon l'endroit où vous obtenez vos données, les autres types d'images que vous rencontrerez très probablement sont les images RGBA, qui permettent la transparence, ou les images en niveaux de gris (luminosité) à canal unique. Téléchargez stinkbug.png sur votre ordinateur pour le reste de ce tutoriel.
Et c'est reparti...
img = mpimg.imread('../../doc/_static/stinkbug.png')
print(img)
[[[0.40784314 0.40784314 0.40784314]
[0.40784314 0.40784314 0.40784314]
[0.40784314 0.40784314 0.40784314]
...
[0.42745098 0.42745098 0.42745098]
[0.42745098 0.42745098 0.42745098]
[0.42745098 0.42745098 0.42745098]]
[[0.4117647 0.4117647 0.4117647 ]
[0.4117647 0.4117647 0.4117647 ]
[0.4117647 0.4117647 0.4117647 ]
...
[0.42745098 0.42745098 0.42745098]
[0.42745098 0.42745098 0.42745098]
[0.42745098 0.42745098 0.42745098]]
[[0.41960785 0.41960785 0.41960785]
[0.41568628 0.41568628 0.41568628]
[0.41568628 0.41568628 0.41568628]
...
[0.43137255 0.43137255 0.43137255]
[0.43137255 0.43137255 0.43137255]
[0.43137255 0.43137255 0.43137255]]
...
[[0.4392157 0.4392157 0.4392157 ]
[0.43529412 0.43529412 0.43529412]
[0.43137255 0.43137255 0.43137255]
...
[0.45490196 0.45490196 0.45490196]
[0.4509804 0.4509804 0.4509804 ]
[0.4509804 0.4509804 0.4509804 ]]
[[0.44313726 0.44313726 0.44313726]
[0.44313726 0.44313726 0.44313726]
[0.4392157 0.4392157 0.4392157 ]
...
[0.4509804 0.4509804 0.4509804 ]
[0.44705883 0.44705883 0.44705883]
[0.44705883 0.44705883 0.44705883]]
[[0.44313726 0.44313726 0.44313726]
[0.4509804 0.4509804 0.4509804 ]
[0.4509804 0.4509804 0.4509804 ]
...
[0.44705883 0.44705883 0.44705883]
[0.44705883 0.44705883 0.44705883]
[0.44313726 0.44313726 0.44313726]]]
Notez le dtype ici - float32. Matplotlib a redimensionné les données 8 bits de chaque canal en données à virgule flottante entre 0,0 et 1,0. En passant, le seul type de données avec lequel Pillow peut fonctionner est uint8. Le traçage Matplotlib peut gérer float32 et uint8, mais la lecture/écriture d'images pour tout format autre que PNG est limitée aux données uint8. Pourquoi 8 bits ? La plupart des écrans ne peuvent restituer que 8 bits par canal de dégradé de couleurs. Pourquoi ne peuvent-ils rendre que 8 bits/canal ? Parce que c'est à peu près tout ce que l'œil humain peut voir. Plus ici (du point de vue de la photographie) : didacticiel sur la profondeur de bits du paysage lumineux .
Chaque liste interne représente un pixel. Ici, avec une image RVB, il y a 3 valeurs. Comme il s'agit d'une image en noir et blanc, R, G et B sont tous similaires. Un RGBA (où A est alpha, ou transparence), a 4 valeurs par liste interne, et une simple image de luminance n'a qu'une valeur (et n'est donc qu'un tableau 2-D, pas un tableau 3-D). Pour les images RVB et RVBA, Matplotlib prend en charge les types de données float32 et uint8. Pour les niveaux de gris, Matplotlib ne prend en charge que float32. Si les données de votre tableau ne répondent pas à l'une de ces descriptions, vous devez les redimensionner.
Tracer des tableaux numpy sous forme d'images #
Ainsi, vous avez vos données dans un tableau numpy (soit en l'important, soit en le générant). Rendons-le. Dans Matplotlib, cela est effectué à l'aide de la imshow()fonction. Ici, nous allons saisir l'objet plot. Cet objet vous permet de manipuler facilement le tracé à partir de l'invite.
imgplot = plt.imshow(img)
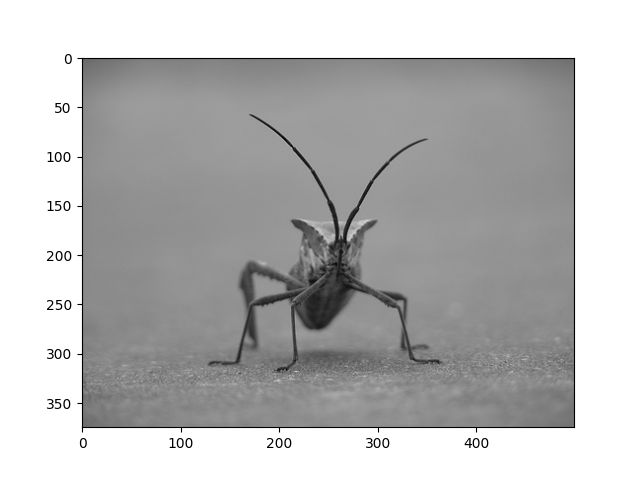
Vous pouvez également tracer n'importe quel tableau numpy.
Application de schémas de pseudo-couleurs aux tracés d'image #
La pseudo-couleur peut être un outil utile pour améliorer le contraste et visualiser plus facilement vos données. Ceci est particulièrement utile lorsque vous faites des présentations de vos données à l'aide de projecteurs - leur contraste est généralement assez faible.
La pseudo-couleur n'est pertinente que pour les images à un seul canal, en niveaux de gris et en luminosité. Nous avons actuellement une image RVB. Étant donné que R, G et B sont tous similaires (voir par vous-même ci-dessus ou dans vos données), nous pouvons simplement choisir un canal de nos données :
lum_img = img[:, :, 0]
# This is array slicing. You can read more in the `Numpy tutorial
# <https://numpy.org/doc/stable/user/quickstart.html>`_.
plt.imshow(lum_img)
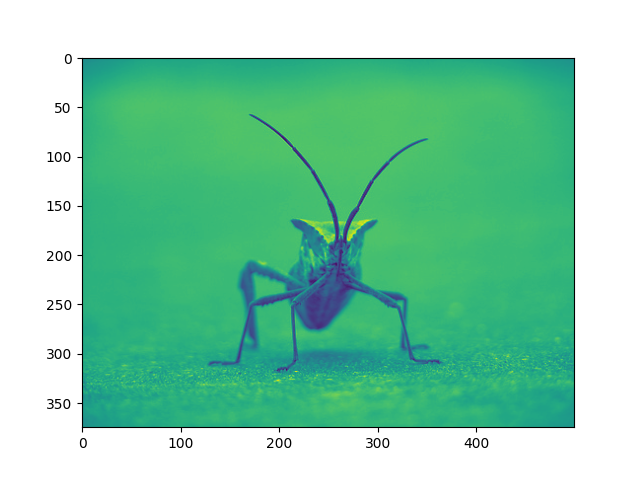
<matplotlib.image.AxesImage object at 0x7f2cdd608610>
Maintenant, avec une image de luminosité (2D, sans couleur), la palette de couleurs par défaut (alias table de consultation, LUT) est appliquée. La valeur par défaut s'appelle viridis. Il y en a beaucoup d'autres parmi lesquels choisir.
plt.imshow(lum_img, cmap="hot")
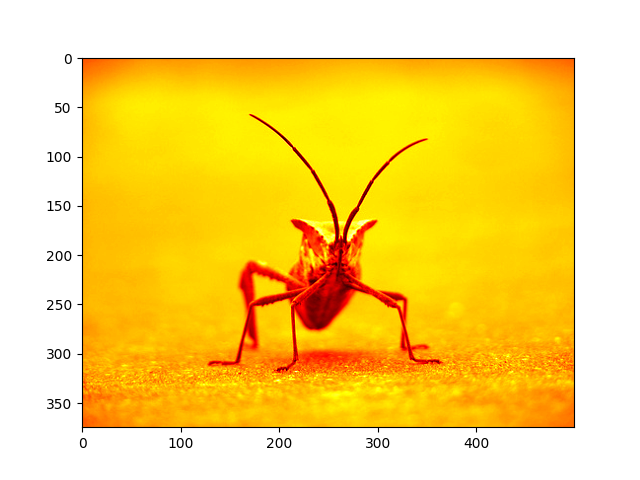
<matplotlib.image.AxesImage object at 0x7f2cddcc2aa0>
Notez que vous pouvez également modifier les palettes de couleurs sur des objets de tracé existants à l'aide de la
set_cmap()méthode :
imgplot = plt.imshow(lum_img)
imgplot.set_cmap('nipy_spectral')
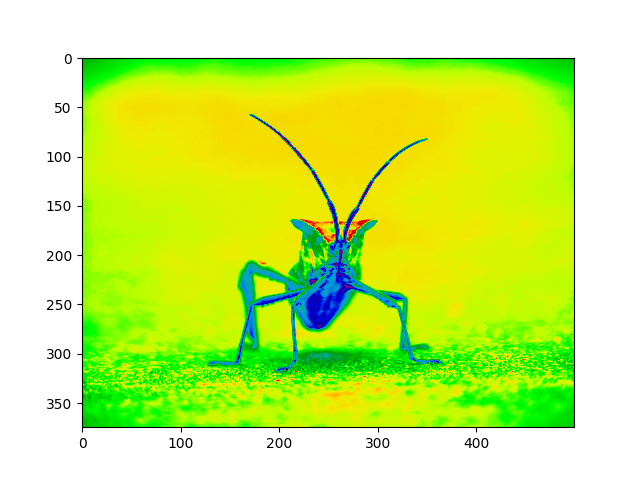
Noter
Cependant, n'oubliez pas que dans le Jupyter Notebook avec le backend en ligne, vous ne pouvez pas apporter de modifications aux tracés qui ont déjà été rendus. Si vous créez imgplot ici dans une cellule, vous ne pouvez pas appeler set_cmap() dessus dans une cellule ultérieure et vous attendre à ce que le tracé précédent change. Assurez-vous que vous entrez ces commandes ensemble dans une cellule. Les commandes plt ne modifieront pas les tracés des cellules précédentes.
Il existe de nombreux autres schémas de palettes de couleurs disponibles. Voir la liste et les images des palettes de couleurs .
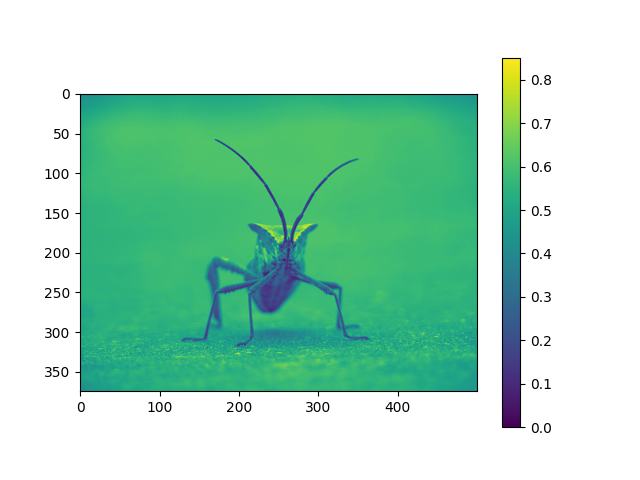
Référence de l'échelle de couleurs #
Il est utile d'avoir une idée de la valeur qu'une couleur représente. Nous pouvons le faire en ajoutant une barre de couleur à votre silhouette :
<matplotlib.colorbar.Colorbar object at 0x7f2cdf5297e0>
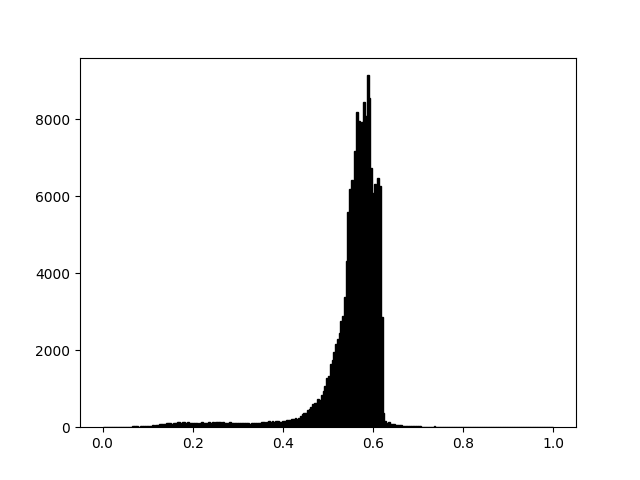
Examen d'une plage de données spécifique #
Parfois, vous souhaitez améliorer le contraste de votre image ou augmenter le contraste dans une région particulière tout en sacrifiant les détails dans des couleurs qui ne varient pas beaucoup ou qui n'ont pas d'importance. Un bon outil pour trouver des régions intéressantes est l'histogramme. Pour créer un histogramme de nos données d'image, nous utilisons la hist()fonction.
(array([2.000e+00, 2.000e+00, 3.000e+00, 3.000e+00, 2.000e+00, 2.000e+00,
3.000e+00, 1.000e+00, 7.000e+00, 9.000e+00, 7.000e+00, 2.000e+00,
7.000e+00, 1.000e+01, 1.100e+01, 1.500e+01, 1.400e+01, 2.700e+01,
2.100e+01, 2.400e+01, 1.400e+01, 3.100e+01, 2.900e+01, 2.800e+01,
2.400e+01, 2.400e+01, 4.000e+01, 2.600e+01, 5.200e+01, 3.900e+01,
5.700e+01, 4.600e+01, 8.400e+01, 7.600e+01, 8.900e+01, 8.000e+01,
1.060e+02, 1.130e+02, 1.120e+02, 9.000e+01, 1.160e+02, 1.090e+02,
1.270e+02, 1.350e+02, 9.800e+01, 1.310e+02, 1.230e+02, 1.110e+02,
1.230e+02, 1.160e+02, 1.010e+02, 1.170e+02, 1.000e+02, 1.010e+02,
9.000e+01, 1.060e+02, 1.260e+02, 1.040e+02, 1.070e+02, 1.110e+02,
1.380e+02, 1.000e+02, 1.340e+02, 1.210e+02, 1.400e+02, 1.320e+02,
1.390e+02, 1.160e+02, 1.330e+02, 1.180e+02, 1.080e+02, 1.170e+02,
1.280e+02, 1.200e+02, 1.210e+02, 1.100e+02, 1.160e+02, 1.180e+02,
9.700e+01, 9.700e+01, 1.140e+02, 1.070e+02, 1.170e+02, 8.700e+01,
1.070e+02, 9.800e+01, 1.040e+02, 1.120e+02, 1.110e+02, 1.180e+02,
1.240e+02, 1.340e+02, 1.200e+02, 1.410e+02, 1.520e+02, 1.360e+02,
1.610e+02, 1.380e+02, 1.620e+02, 1.570e+02, 1.350e+02, 1.470e+02,
1.690e+02, 1.710e+02, 1.820e+02, 1.980e+02, 1.970e+02, 2.060e+02,
2.160e+02, 2.460e+02, 2.210e+02, 2.520e+02, 2.890e+02, 3.450e+02,
3.620e+02, 3.760e+02, 4.480e+02, 4.630e+02, 5.170e+02, 6.000e+02,
6.200e+02, 6.410e+02, 7.440e+02, 7.120e+02, 8.330e+02, 9.290e+02,
1.061e+03, 1.280e+03, 1.340e+03, 1.638e+03, 1.740e+03, 1.953e+03,
2.151e+03, 2.290e+03, 2.440e+03, 2.758e+03, 2.896e+03, 3.384e+03,
4.332e+03, 5.584e+03, 6.197e+03, 6.422e+03, 6.404e+03, 7.181e+03,
8.196e+03, 7.968e+03, 7.474e+03, 7.926e+03, 8.460e+03, 8.091e+03,
9.148e+03, 8.563e+03, 6.747e+03, 6.074e+03, 6.328e+03, 5.291e+03,
6.472e+03, 6.268e+03, 2.864e+03, 3.760e+02, 1.620e+02, 1.180e+02,
1.270e+02, 9.500e+01, 7.600e+01, 8.200e+01, 6.200e+01, 6.700e+01,
5.600e+01, 5.900e+01, 4.000e+01, 4.200e+01, 3.000e+01, 3.400e+01,
3.200e+01, 4.300e+01, 4.200e+01, 2.300e+01, 2.800e+01, 1.900e+01,
2.200e+01, 1.600e+01, 1.200e+01, 1.800e+01, 9.000e+00, 1.000e+01,
1.700e+01, 5.000e+00, 2.100e+01, 1.300e+01, 8.000e+00, 1.200e+01,
1.000e+01, 8.000e+00, 8.000e+00, 5.000e+00, 1.300e+01, 6.000e+00,
3.000e+00, 7.000e+00, 6.000e+00, 2.000e+00, 1.000e+00, 5.000e+00,
3.000e+00, 3.000e+00, 1.000e+00, 1.000e+00, 1.000e+00, 5.000e+00,
0.000e+00, 1.000e+00, 3.000e+00, 0.000e+00, 1.000e+00, 1.000e+00,
2.000e+00, 1.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00,
0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00,
0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00,
0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00,
0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00,
0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00,
0.000e+00, 0.000e+00, 0.000e+00, 0.000e+00]), array([0. , 0.00390625, 0.0078125 , 0.01171875, 0.015625 ,
0.01953125, 0.0234375 , 0.02734375, 0.03125 , 0.03515625,
0.0390625 , 0.04296875, 0.046875 , 0.05078125, 0.0546875 ,
0.05859375, 0.0625 , 0.06640625, 0.0703125 , 0.07421875,
0.078125 , 0.08203125, 0.0859375 , 0.08984375, 0.09375 ,
0.09765625, 0.1015625 , 0.10546875, 0.109375 , 0.11328125,
0.1171875 , 0.12109375, 0.125 , 0.12890625, 0.1328125 ,
0.13671875, 0.140625 , 0.14453125, 0.1484375 , 0.15234375,
0.15625 , 0.16015625, 0.1640625 , 0.16796875, 0.171875 ,
0.17578125, 0.1796875 , 0.18359375, 0.1875 , 0.19140625,
0.1953125 , 0.19921875, 0.203125 , 0.20703125, 0.2109375 ,
0.21484375, 0.21875 , 0.22265625, 0.2265625 , 0.23046875,
0.234375 , 0.23828125, 0.2421875 , 0.24609375, 0.25 ,
0.25390625, 0.2578125 , 0.26171875, 0.265625 , 0.26953125,
0.2734375 , 0.27734375, 0.28125 , 0.28515625, 0.2890625 ,
0.29296875, 0.296875 , 0.30078125, 0.3046875 , 0.30859375,
0.3125 , 0.31640625, 0.3203125 , 0.32421875, 0.328125 ,
0.33203125, 0.3359375 , 0.33984375, 0.34375 , 0.34765625,
0.3515625 , 0.35546875, 0.359375 , 0.36328125, 0.3671875 ,
0.37109375, 0.375 , 0.37890625, 0.3828125 , 0.38671875,
0.390625 , 0.39453125, 0.3984375 , 0.40234375, 0.40625 ,
0.41015625, 0.4140625 , 0.41796875, 0.421875 , 0.42578125,
0.4296875 , 0.43359375, 0.4375 , 0.44140625, 0.4453125 ,
0.44921875, 0.453125 , 0.45703125, 0.4609375 , 0.46484375,
0.46875 , 0.47265625, 0.4765625 , 0.48046875, 0.484375 ,
0.48828125, 0.4921875 , 0.49609375, 0.5 , 0.50390625,
0.5078125 , 0.51171875, 0.515625 , 0.51953125, 0.5234375 ,
0.52734375, 0.53125 , 0.53515625, 0.5390625 , 0.54296875,
0.546875 , 0.55078125, 0.5546875 , 0.55859375, 0.5625 ,
0.56640625, 0.5703125 , 0.57421875, 0.578125 , 0.58203125,
0.5859375 , 0.58984375, 0.59375 , 0.59765625, 0.6015625 ,
0.60546875, 0.609375 , 0.61328125, 0.6171875 , 0.62109375,
0.625 , 0.62890625, 0.6328125 , 0.63671875, 0.640625 ,
0.64453125, 0.6484375 , 0.65234375, 0.65625 , 0.66015625,
0.6640625 , 0.66796875, 0.671875 , 0.67578125, 0.6796875 ,
0.68359375, 0.6875 , 0.69140625, 0.6953125 , 0.69921875,
0.703125 , 0.70703125, 0.7109375 , 0.71484375, 0.71875 ,
0.72265625, 0.7265625 , 0.73046875, 0.734375 , 0.73828125,
0.7421875 , 0.74609375, 0.75 , 0.75390625, 0.7578125 ,
0.76171875, 0.765625 , 0.76953125, 0.7734375 , 0.77734375,
0.78125 , 0.78515625, 0.7890625 , 0.79296875, 0.796875 ,
0.80078125, 0.8046875 , 0.80859375, 0.8125 , 0.81640625,
0.8203125 , 0.82421875, 0.828125 , 0.83203125, 0.8359375 ,
0.83984375, 0.84375 , 0.84765625, 0.8515625 , 0.85546875,
0.859375 , 0.86328125, 0.8671875 , 0.87109375, 0.875 ,
0.87890625, 0.8828125 , 0.88671875, 0.890625 , 0.89453125,
0.8984375 , 0.90234375, 0.90625 , 0.91015625, 0.9140625 ,
0.91796875, 0.921875 , 0.92578125, 0.9296875 , 0.93359375,
0.9375 , 0.94140625, 0.9453125 , 0.94921875, 0.953125 ,
0.95703125, 0.9609375 , 0.96484375, 0.96875 , 0.97265625,
0.9765625 , 0.98046875, 0.984375 , 0.98828125, 0.9921875 ,
0.99609375, 1. ]), <BarContainer object of 256 artists>)
Le plus souvent, la partie "intéressante" de l'image se situe autour du pic, et vous pouvez obtenir un contraste supplémentaire en coupant les régions au-dessus et/ou en dessous du pic. Dans notre histogramme, il semble qu'il n'y ait pas beaucoup d'informations utiles dans le haut de gamme (pas beaucoup de choses blanches dans l'image). Ajustons la limite supérieure, de manière à "zoomer" efficacement sur une partie de l'histogramme. Pour ce faire, nous passons l'argument clim à imshow. Vous pouvez également le faire en appelant la
set_clim()méthode de l'objet image plot, mais assurez-vous de le faire dans la même cellule que votre commande plot lorsque vous travaillez avec Jupyter Notebook - cela ne changera pas les tracés des cellules précédentes.
Vous pouvez spécifier la clim dans l'appel à plot.
imgplot = plt.imshow(lum_img, clim=(0.0, 0.7))
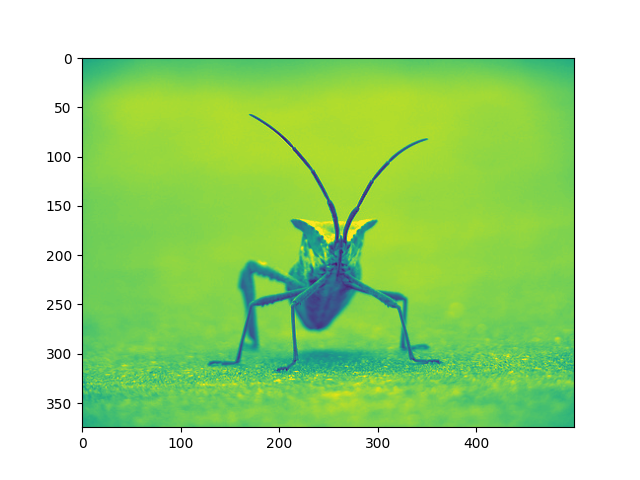
Vous pouvez également spécifier la clim en utilisant l'objet retourné
fig = plt.figure()
ax = fig.add_subplot(1, 2, 1)
imgplot = plt.imshow(lum_img)
ax.set_title('Before')
plt.colorbar(ticks=[0.1, 0.3, 0.5, 0.7], orientation='horizontal')
ax = fig.add_subplot(1, 2, 2)
imgplot = plt.imshow(lum_img)
imgplot.set_clim(0.0, 0.7)
ax.set_title('After')
plt.colorbar(ticks=[0.1, 0.3, 0.5, 0.7], orientation='horizontal')
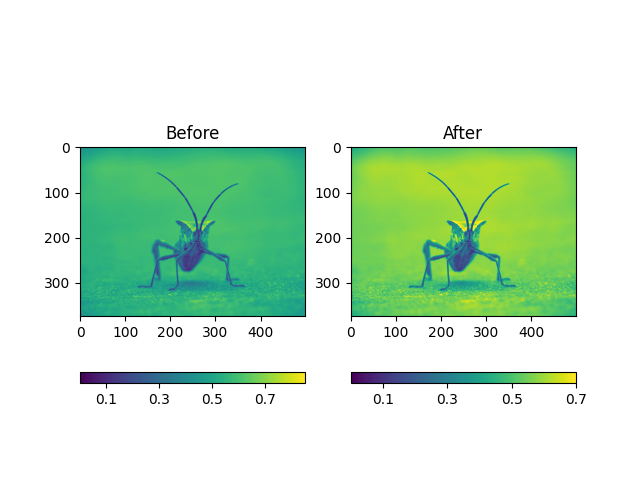
<matplotlib.colorbar.Colorbar object at 0x7f2cdf75fa30>
Schémas d'interpolation de tableau #
L'interpolation calcule ce que la couleur ou la valeur d'un pixel "devrait" être, selon différents schémas mathématiques. Cela se produit généralement lorsque vous redimensionnez une image. Le nombre de pixels change, mais vous voulez les mêmes informations. Comme les pixels sont discrets, il manque de l'espace. L'interpolation est la façon dont vous remplissez cet espace. C'est pourquoi vos images paraissent parfois pixélisées lorsque vous les agrandissez. L'effet est plus prononcé lorsque la différence entre l'image d'origine et l'image agrandie est plus grande. Prenons notre image et réduisons-la. Nous supprimons efficacement les pixels, en n'en gardant que quelques-uns. Maintenant, lorsque nous les traçons, ces données sont agrandies à la taille de votre écran. Les anciens pixels ne sont plus là et l'ordinateur doit dessiner des pixels pour remplir cet espace.
Nous utiliserons la bibliothèque Pillow que nous avons utilisée pour charger l'image également pour redimensionner l'image.
from PIL import Image
img = Image.open('../../doc/_static/stinkbug.png')
img.thumbnail((64, 64)) # resizes image in-place
imgplot = plt.imshow(img)
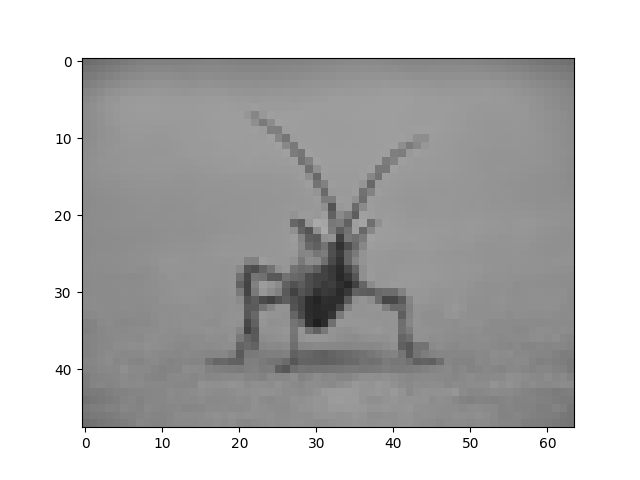
Nous avons ici l'interpolation par défaut, bilinéaire, puisque nous n'avons donné imshow()aucun argument d'interpolation.
Essayons-en d'autres. Voici "le plus proche", qui ne fait aucune interpolation.
imgplot = plt.imshow(img, interpolation="nearest")
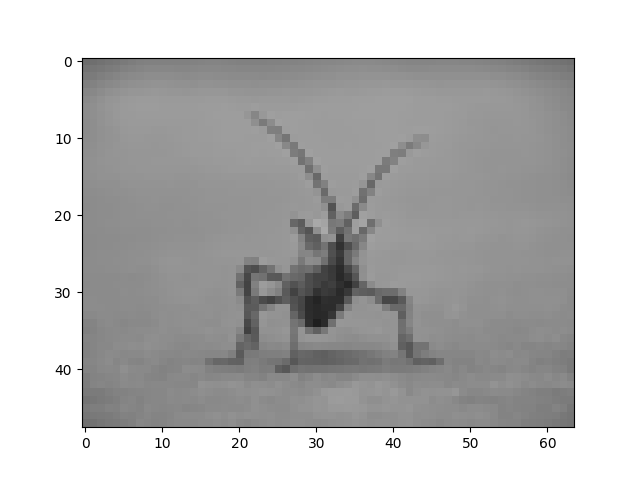
et bicubique :
imgplot = plt.imshow(img, interpolation="bicubic")
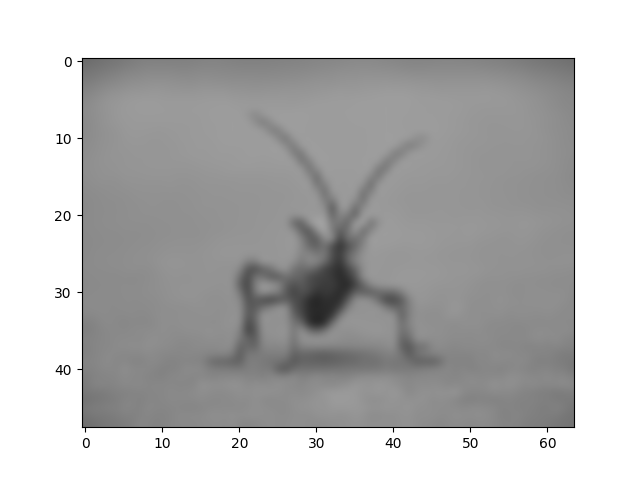
L'interpolation bicubique est souvent utilisée lors de l'agrandissement de photos - les gens ont tendance à préférer le flou au pixélisé.
Durée totale d'exécution du script : (0 minutes 8,344 secondes)